鄭明月團隊揭示AI模型在胰腺癌藥物研發(fā)中的應用
2024年6月25日,中國科學院上海藥物研究所原創(chuàng)新藥研究全國重點實驗室鄭明月團隊在《Nature Communications》雜志發(fā)表了題為"Deep representation learning of chemical-induced transcriptional profile for phenotype-based drug discovery"的研究論文,該團隊認為�,將TranSiGen整合到藥物研發(fā)中�����,對于推進生物醫(yī)學具有重大前景。
TranSiGen 可分析基底細胞基因表達和分子結構�,以高精度重建化學誘導的轉錄譜����。通過捕獲細胞和化合物信息,TranSiGen 衍生的表征在各種下游任務中表現(xiàn)出療效����,例如,基于配體的虛擬篩選�、藥物反應預測和基于表型的藥物再利用。值得注意的是���,TranSiGen在胰腺癌藥物研發(fā)中的應用��,凸顯了其識別有效化合物的潛力��。
研究背景
藥物研發(fā)領域正在經歷由人工智能 (AI) 驅動的革新��。雖然基于靶點的方法����,長期以來一直主導著該領域,但它們的局限性:包括缺乏明確的靶點���、脫靶效應和不太令人滿意的治療反饋����,推動了基于表型的方法的興起�����。這些方法側重于對候選藥物的全面細胞反應�,提供對疾病機制更全面的理解,并可能揭示新的藥物靶點和治療途徑�。
轉錄組學數(shù)據(jù)分析在藥物研發(fā)和理解疾病機制方面,起著至關重要的作用���。通過捕捉不同生物學背景下的全球基因表達情況�,它提供了對細胞和生物狀態(tài)的豐富見解����。高通量RNA測序(RNA-seq)技術,促進了大規(guī)?;虮磉_圖譜的生成,這些基因表達圖譜為細胞如何對各種破壞做出反應���,提供了寶貴的信息��。對這些特征的探索���,在藥物研發(fā)中起著核心作用�,有助于闡明藥物的作用機制 (MOA)���。
盡管基因表達圖譜具有巨大價值,但類藥物分子和細胞系的組合復雜性��,限制了通過高通量實驗進行詳盡的探索�����。這一挑戰(zhàn)加速了深度學習模型的發(fā)展�,該模型能夠使用公開數(shù)據(jù)預測新型化學品的轉錄譜。DLEPS 是一種深度神經網絡����,旨在預測對新化學物質的基因表達反應,而沒有細胞類型特異性�。此外,DeepCE14和 CIGER15利用單熱編碼來區(qū)分細胞類型�,從不同的圖譜中進行學習�。MultiDCP通過結合細胞背景來預測環(huán)境依賴性基因表達和細胞活力��,從而能夠對新型細胞系�,進行特定環(huán)境的預測。
然而���,直接擬合基因表達值的監(jiān)督學習模型�����,可能難以將真正的擾動信號與混雜因素����,以及表達譜中的固有噪聲區(qū)分開來��。為了解決數(shù)據(jù)的局限性����,并生成新的圖譜,本研究提出了TranSiGen����,這是一個基于VAE的框架,利用自監(jiān)督學習�����,來降噪和重建轉錄圖譜,從而能夠推斷新圖譜的模型��。TranSiGen同時學習3個關鍵分布:無擾動的基礎分布�、化學誘導的擾動分布,以及它們之間的映射關系����。這種自監(jiān)督學習的方法,有效地減輕了數(shù)據(jù)中的噪聲����,并揭示了潛在的擾動信號�����。TranSiGen 具有幾個關鍵優(yōu)勢:(1) 改進轉錄譜的推斷:通過與基線模型的比較�,證明了 TranSiGen在推斷基礎譜、化學擾動譜和相應的差異表達基因 (DEG) 方面的卓越性能�。(2)細胞和化合物特征的統(tǒng)一表示:TranSiGen生成的擾動圖譜有效地捕獲了細胞和化合物特征,這可以通過區(qū)分細胞系和藥物MOA的可視化分析來證明��。(3) 在下游任務中的多功能應用:TranSiGen 衍生的表征已被證明在各種任務中有效��,包括基于配體的虛擬篩選、藥物反應預測和基于表型的藥物再利用�。它在篩選胰腺癌化合物中的應用,以及隨后的體外驗證和高命中率�,證明了TranSiGen基于表型的方法,在識別有效化合物方面的強大功能��。重要的是����,TranSiGen與基于表型的藥物研發(fā)管線的整合,有望顯著提高研發(fā)效率并降低成本�����。
研究進展
基于表型的藥物再利用治療胰腺癌
將化學誘導的轉錄譜與疾病相關聯(lián)��,有助于確定治療特定疾病的潛在化合物���。TranSiGen衍生的轉錄譜�����,可以與來自化學處理和未處理疾病狀態(tài)的譜一起使用�,以篩選用于疾病治療的候選化合物��。
在本研究中,團隊將TranSiGen整合到基于表型的胰腺癌藥物再利用管線中����,評估其從 PRISM Reutilposing 數(shù)據(jù)集的 1,625 種化合物池中,優(yōu)先篩選 YAPC 胰腺癌細胞系敏感化合物的能力�����。團隊使用了兩種基于表型的策略��,并將它們與傳統(tǒng)的基于結構相似性的方案進行了比較�。TranSiGen_DRUG使用已批準的胰腺癌藥物的真實DEG,來識別具有類似擾動效應的化合物�����。相反�,TranSiGen_DISEASE尋找可以逆轉胰腺癌DEGs的化合物�����。
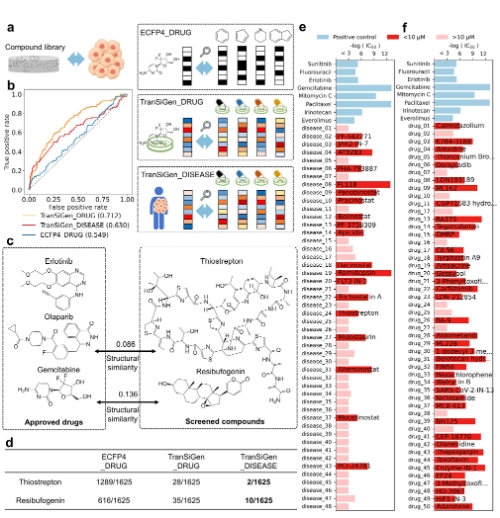
三種方法的篩選性能如下圖所示��。ECFP4_DRUG 產生較差的預測分類性能�,其次是 TranSiGen_DISEASE�,較好的是TranSiGen_DRUG�����。值得注意的是��,TranSiGen_DISEASE方法不需要任何化學處理的配置文件����,模擬疾病缺乏已知治療藥物的場景。這是基于結構相似性的策略��,無法解決的挑戰(zhàn)��。
總體而言�����,TranSiGen 擴展了可通過預測的擾動曲線進行篩選的化合物范圍��。它可以很容易地集成到基于表型的藥物再利用管線中�,從而提高藥物研發(fā)效率并較大限度地降低成本。
a 藥物再利用戰(zhàn)略的流程圖���。b 基于表型的策略和基于結構相似性的策略的篩選性能����。C TranSiGen_DISEASE篩選出能夠抑制胰腺癌細胞的化合物,以及它們與已批準藥物的較大結構相似性����。d 不同篩選策略中硫代霉素和resibufogenin的排名。e 通過TranSiGen_DISEASE篩選的前 50 種化合物���,及其各自的細胞增殖抑制活性�。f 通過TranSiGen_DRUG篩選的前50種化合物�,及其相應的細胞增殖抑制活性。
研究結論
本研究證明�,TranSiGen 在推斷基礎剖面、化學誘導的擾動剖面和相應的 DEG 方面�,優(yōu)于現(xiàn)有的模型。此功能為擴展和增強現(xiàn)有藥物研發(fā)數(shù)據(jù)集�����,開辟了新的途徑����。TranSiGen的核心優(yōu)勢,在于它能夠克服基因表達譜中固有的干擾和混雜因素����,提供一種標準化的方法,來表征與細胞背景和化合物效應相關的表型信息��。這種標準化有助于各種下游任務的集成和效率提高����,包括基于配體的虛擬篩選、藥物反應預測和基于表型的藥物再利用���。值得注意的是��,它在基于表型的胰腺癌藥物再利用中的應用��,以及隨后的體外驗證���,展示了其在真實世界藥物研發(fā)場景中的前景。
TranSiGen為藥物研發(fā)中�����,基于VAE的模型和自監(jiān)督學習方法的持續(xù)探索�,奠定了基礎。團隊未來的工作,將集中在解決TranSiGen中不同來源的數(shù)據(jù)的異質性���,并增強模型相對于其他平臺的基礎配置文件的泛化性能���,以拓寬其應用領域。此外�,團隊計劃通過整合之前的生物學知識(例如,通路和基因本體)���,來提高模型的精確度和可解釋性�。除了目前在藥物研發(fā)中的應用����,團隊還渴望研究TranSiGen在精準醫(yī)學和疾病建模方面的潛在作用,并揭示這些領域的巨大前景����。該領域的終目標,是創(chuàng)建一個真正全面的框架�����,以有效利用高維基因表達數(shù)據(jù)��。這將加速藥物研發(fā),并揭示疾病機制的復雜性�。TranSiGen憑借其獨特的優(yōu)勢和可擴展性�,標志著朝著實現(xiàn)這一目標,邁出了寶貴的一步��。
聲明:本文版權歸原作者所有����,轉載文章僅為傳播更多信息,如作者信息標記有誤���,或侵犯您的版權�,請聯(lián)系我們����,我們將在及時修改或刪除內容,聯(lián)系郵箱:marketing@360worldcare.com